Alfa sarmal
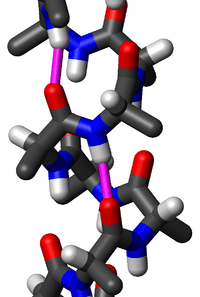
Protein ikincil yapısında yaygın bir motif olan alfa sarmal (α-sarmal), sağ-elli burgulu bir biçimdir, omurgadaki her bir N-H grubu, kendinden dört amino asit kalıntısı gerideki omurgadaki C=O grubuna bir hidrojen bağı verir ( hidrojen bağlanması). Bu ikincil yapı bazen klasik Pauling-Corey-Branson alfa sarmalı olarak da adlandırılır (aşağıdaki metne bakınız). Proteinlerin lokal yapı tipleri arasında α-sarmal, en düzenli olan, diziden öngörüsü yapılması en kolay olan ve ayrıca en yaygın olandır.
Tarihsel gelişim
1930'larda William Astbury, nemli yün veya saç lifleri gerildikleri zaman X-ışını lif kırınımında büyük değişiklikler meydana geldiğini göstermiştir. Verilerden anlaşılan, gerilmemiş liflerin 5,1 Å'luk bir tekrarlı birime sahip burgulu bir molekül yapısı olduğuydu.
Astbury, lifler için önce dirsekli bir yapı öne sürdü. Daha sonra, diğer araştırmacılara (özellikle Amerikalı kimyacı Maurice Higgins'e) katılıp aşağıdaki görüşleri savundu:
- Gerilmemiş protein molekülleri bir sarmak oluşturmaktadır (bunu α-biçim olarak adlandırdı); ve
- Gerilme sarmalın açılmasına neden olmakta ve uzamış bir hâl oluşturmaktadır (bunu β-biçim olarak adlandırdı).
Ayrıntılarında hatalı olmasına rağmen, Astbury'nin bu biçimler hakkındaki modelleri esas olarak doğruydu ve 1951'de Linus Pauling, Robert Corey ve Herman Branson tarafından geliştirilen, ikincil yapının modern elemanları olan α-sarmal ve β-ipliğe karşılık gelmektedir (Astbury'nin özgün adlandırması korunmuştur). Bu araştırmacıların makalesinde hem sağ-elli hem de sol-elli sarmalların varlığın göstermiş, ama 1960'ta yayımlanan miyoglobin kristal yapısı[1] sağ-elli biçimin yaygın olduğunu göstermiştir. Hans Neurath Astbury'nin modelinin ayrıntılarda doğru olamayacağını göstermiştir, çünkü modeldeki atomların birbirine temas etmesi gerekmektedir.[2] Neurath'ın makalesi ve Astbury'nin verileri H. S. Taylor,[3] Maurice Huggins[4] ve Bragg ve çalışma arkadaşlarını[5] keratin için bir yapı önermelerine ilham kaynağı olmuştur, bu önerilen yapı modern α-sarmala benzemektedir.
Modern α-sarmalın modellenmesinde iki anahtar gelişme, (1) amino asitler ve peptitlerin kristal yapılarının çözülmesi ve Pauling'in düzlemsel peptit bağının öngörüsü sayesinde bağ geometrisinin belirlenmesi, ve (2) onun, sarmaldaki bir dönmede bulunan kalıntı sayısı konusundaki varsayımından vazgeçmesi oldu. Tarihsel dönüm noktası 1948 ilkbaharı başlarında oldu, Pauling nezle oldu ve yatağına girdi. Canı sıkıldığından ötürü, boyutları yaklaşık doğru olan bir peptit zincirini bir kağıt şerit üzerine çizdi ve onu bir sarmal şeklinde katladı, düzlemsel peptit bağlarını korumaya dikkat ederek. Birkaç denemeden sonra, fiziksel olarak makul hidrojen bağları olan bir model meydana getirdi. Pauling sonra modelini yayımlamadan önce doğrulamak için Corey ve Branson ile çalıştı.[6] 1954'te Pauling "kimyasal bağ üzerindeki araştırması ve bunun karmaşık bileşiklerin yapılarının çözülmesine uygulanması" için ilk Nobel Ödülünü kazandı[7]
Yapı
Geometri ve hidrojen bağlanması
α sarmaldaki amino asitler sağ-elli sarmal yapıya sahiptir, her amino asit kalıntısı sarmal üzerinde 100°'lik bir dönmeye (yani sarmalda bir tam dönme başına 3,6 kalıntı vardır) ve sarmal ekseni boyunca 1,5 Å'luk bir ötelenmeye karşılık gelir. (Çok miktarda kiral olmayan glisin amino asit içeriği olunca kısa sol-elli sarmal parçaları bazen oluşabilir, ama diğer normal, biyolojik L-amino asitler) için bunlar uygun değildir.) Alfa sarmalın hatvesi (sarmalın bir tam dönüşü için eksen boyunca olan uzaklık) 5,4 Å'dur, bu 1,5 ve 3,6 sayılarının çarpımıdır. En önemli olan, bir amino asit kalıntısındaki N-H grubunun kendinden dört kalıntı gerideki C=O grubu ile bir hidrojen bağı kurmasıdır; bu tekrarlayan hidrojen bağlanması α sarmalın en belirgin özelliğidir. Resmî uluslararası adlandırma kuralları[8] α sarmalların tanımlanması için iki yol belirtir: kural 6.2, tekrar eden φ,ψ torsiyon açılarına göre tanımlar (metinde aşağıya bakınız), kural 6.3 ise hatve ve hidrojen bağlanmasının oluşturduğu birleşik örüntüye göre tanımlar. Protein yapılarında yer alan alfa sarmallar çeşitli berimsel yöntemlerle tespit edilebilir, bunlardan biri DSSP (Dictionary of Protein Secondary structure, Protein ikincil yapı sözlüğü)'dür.[9]
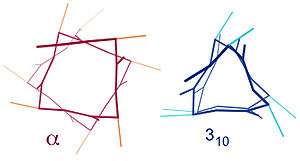
Benzer yapılar arasında 310 sarmalı ( hidrojen bağlanmalı) ve π-sarmal ( hidrojen bağı) sayılabilir. α sarmal 3.613 sarmal olarak da tanımlanabilir çünkü, daha sıkı olan 310 sarmala kıyasla, i + 4 aralıklama H-bağlı halkaya fazladan 3 atom daha ekler. Alt yazılı endeksler hidrojen bağı ile meydana gelen kapalı halkadaki (hidrojen atomu da dahil olmak üzere) atom sayısına karşılık gelir.[10]
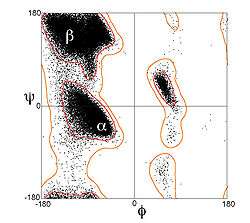
α-sarmallardaki kalıntılar tipik olarak (-60°, -45°) civarında omurga (φ, ψ) dihedral açıları oluştururlar. Daha genel ifade etmek gerekirse, bir kalıntının ψ dihedral açısı ile sonraki kalıntının φ dihedral açısının toplamı yaklaşık -105°'ye eşitliğini sağlayacak dihedral açılar oluştururlar. Bunun bir sonucu olarak, α-sarmal dihedral açıları, genelde Ramachandran çiziminde (-90°, -15°) ile (-35°, -70°) arasında çaprak bir şerit üzerine düşerler. Buna karşın, 310 sarmallar için dihedral açı toplamları yaklaşık -75°, π-sarmal için ise yaklaşık -130°'dir. Trans izomerlerden oluşan herhangi bir polipeptit sarmal için kalıntı başına dönme açısı Ω, aşağıdaki denklem ile ifade edilir:[12]
α-sarmaldaki atomlar sıkıca istiflenmiştir; sarmal içinde hemen hiç boş hacim yoktur. Amino asit yan zincirleri sarmalın dışında yer alır ve kabaca "aşağıya" (yani N-uca doğru) uzanırlar, bir çam ağacı gibi. Protein yapısını çözerken ön çalışmalardaki düşük çözünürlüklü elektron yoğunluk haritalarına bakılırken bu özellikten yararlanılarak protein omurgasının yönü tayin edilebilir.
Alfa sarmalları temsil etmek için 2B (boyutlu) çizimler
α-sarmalları temsil etmek için iki farklı cins 2 boyutlu çizim kullanılır. Biri "sarmal tekerlek" (helical wheel),[13] öbürü "wenxiang çizimi"dir.[14] İkincisinin adı, Çinde sivrisinekleri kaçırmak için kullanılan spiral şekilli bir tütsüye benzemesinden dolayıdır: 蚊香 (“venşiang” olarak okunur).
Wenxiang çiziminde her amino asit kalıntısı, onun tek harfli koduna karşılık gelen, içinde bir harf olan bir daire ile gösterilir. Hidrofobik bir kalıntı, içi dolu bir daire içinde beyaz bir senbolle belirtilir, hidrofilik kalıntılar içi boş bir daire içinde siyah bir sembolle belirtilir. 2 Boyutlu bir gösterim şekli olarak wenxiang çiziminin, sarmal tekerlek çizimine kıyasla aşağıda belirtilen avantajları vardır: (1) ne kadar uzun olursa olsun, bir alfa sarmaldaki amino asit kalıntılarının konumunu gösterebilmesi; (2) alfa-sarmalın yönünü gösterebilmesi; ve (3) bir α-sarmalı oluşturan amino asit kalıntıları hakkında daha fazla bilgi verebilmesi.[14][15]
Kararlılık
Proteinlerde görülen sarmallar dört ilâ kırk kalıntı uzunluğunda olabilir, ama tipik bir sarmalda yaklaşık 10 amino asit kalıntısı (yaklaşık 3 dönme) bulunur. Genelde kısa polipeptitler çözelti içinde fazla alfa sarmal yapı göstermezler, çünkü proteinin katlanmasıyla ilişkili olan entropik masraf, yeterli miktarda stabilize edici etkileşimlerle telafi edilmez. Genelde, α-sarmalların omurga hidrojen bağları, β-yapraklarda bulunanlardakinden çok daha zayıftır ve ortamdaki su molekülleri tarafından kolaylıkla saldırıya uğrarlar. Ancak, hücre zarı gibi daha hidrofobik ortamlarda, trifloroetanol (TFE) gibi çözücülerin varlığında, veya bir çözücünün bulunmadığı gaz ortamında,[16] oligopeptitler kendiliklerinden stabil, α-sarmallı yapı oluştururlar. Peptitlere çapraz bağlar eklenerek sarmal katlamalar daha stabilize edilebilir. Çapraz bağlar, çözülmüş yapıyı destabilize eder ve sarmal hâl ile rekabet eden yapıları ortadan kaldırır.[17]
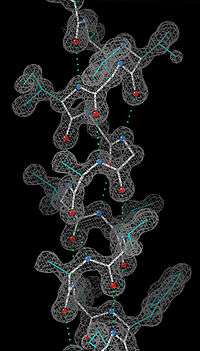
Deneysel tespit
α-sarmal hidrojen bağları ve omurga biçimi ile tanımlı olduğu için α-sarmal yapı için en ayrıntılı deneysel kanıt, bir örneği sağda görülen, atomik çözünürlüklü X ışını kristalografisi ile elde edilmiştir. Omurga karbonil oksijenlerinin aşağıya (C-ucuna doğru) dönük oldukları ama dışa doğru açıldıkları ve H-bağlarının sarmal eksenine yaklaşık paralel oldukları bellidir. NMR spektroskopisi ile çözülen protein yapılarında da sarmallar iyice görünür, bitişik sarmal dönmeleri arasındaki NOE (Nükleer Overhauser Etkisi) bağlantıları gözlemlenebilir. Bazı durumlarda bireysel hidrojen bağları, NMR'de küçük ölçekli kenetleme olarak gözlemlenebilir.
Genel sarmal yapıyı belirlemek için çeşitli düşük çözünürlüklü yöntemler vardır. NMR kimyasal kaymaları (özellikle , ve atomlarının) ve artık dipol bağlaşımları (İng. residual dipole coupling) genelde sarmallar için karakteristiktir. Sarmalların uzak-mor ötesi (170-250 nm) dairesel dikroizm spektrumu da onlara hastır, ~208 nm ve ~222 nm'de belirgin bir çifte minimum gösterir. Kızılötesi spektrometresi ender kullanılır çünkü α-sarmal spektrumu rastgele sarım (İng. random coil) spektrumuna benzerdir (ama bu ikisi hidrojen-döteryum değiştokuşu ile ayırdedilebilir). Kriyo elektron mikroskopisi ile bir protein içindeki bireysel α-sarmalları ayırdedebilir, ama bu yolla amino asit kalıntılarının yerlerinin belirlenmesi hâlâ aktif bir araştırma konusudur.
Amino asitlerin uzun homopolimerleri eğer çözünür iseler genelde sarmallar oluştururlar. Bu uzun, izole sarmallar başka yöntemlerle tespit edilebilir, örneğin dielektrik gevşeme ve difüzyon katsayısı ölçümü ile. Aslında, bu yöntemler kullanılarak tespit edilen özellik, bir sarmalın karakteristik ince ve uzun (prolat, yani puro şekilli) hidrodinamik şekli veya onun büyük dipol momentidir.
Amino asit eğilimleri
Farklı amino asitlerin α-sarmal oluşturmakta farklı eğilimler gösterirler. Lizin, yüksüz glutamat, metyonin, alanin ve lösin amino asit kalıntılarının (tek harfli amino asit koduyla "KEMAL") sarmal oluşturmaya büyük yatkınlıkları vardır, buna karşın prolin ve glisinin sarmal oluşturma yatkınlığı düşüktür.[18] Prolin kalıntısı bir sarmalı ya bozar ya da büker, çünkü hem amit hidrojen bağı oluşturamaz (amit gruubunun hidrojeni olmadığı için), hem de yan zinciri bir evvelki dönmedeki omurgaya sterik olarak müdahale eder. Bu yüzden sarmal ekseninde 30°'lik bir bükülme meydana gelir.[10] Ancak, prolin genelde sarmalın ilk amino asit kalıntısı olarak görülür, muhtemelen neden olduğu yapısal sertlik nedeniyle. Glisin de sarmalı bozar, ama bunun nedeni, prolinin aksine, onun yapısal esnekliğidir; glisinin nispeten kısıtlanmış olan α-sarmal yapıyı benimsemesi entropik olarak masraflıdır.
Dipol momenti
Sarmal ekseni boyunca tüm karbonil grupların bireysel dipol momentlerinin toplu etkisi nedeniyle sarmalın tamamının bir dipol momenti vardır. Bu, entropik etkilerle sarmalın destabilize olmasına neden olabilir. Bunun sonucu olarak, α sarmalların ucu genelde glutamik asit gibi negatif yüklü bir amino asit kalıntısı ile sonlanır, bu sarmal dipolünü nötralize etmek için. Daha ender (ve stabilizasyonda daha az etkili) olarak C-uç, lizin gibi pozitif yüklü bir amino asit kalıntısı ile sonlanır. N-uçtaki (N-terminal) pozitif yük yaygın olarak negatif yüklü ligandların (fosfat grupları gibi) bağlanmasına yarar, bu özellikle etkili bir mekanizmadır çünkü omurgadaki amit grupları hidrojen bağı vericisi görevi yapabilirler.
Daha büyük ölçekli toplanmalar
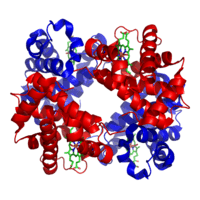
X-ışını kristalografisi ile yapıları çözülmüş ilk iki protein olan Miyoglobin ve hemoglobin, yaklaşık %70 α-sarmal ile benzer katlamalara sahiptir, yapılarının geri kalanı, sarmalları birbirine bağlayan, tekrarlamalı olmayan bölgelerden, veya sarımlardan (İng. coil) oluşur. Proteinleri en belirgin katlanmalarına göre sınıflandıran SCOP (Structural Classification of Proteins) veritabanı, "tüm-α" proteinler için özellikle geniş bir kategori barındırır.
Sarımlı sarım (İng. coiled coil) α sarmalları iki veya daha çok sarmalın birbiri etrafına sarılıp bir "süper sarım" oluşturduğu çok stabil yapılardır. Sarımlı sarımlar, yedili tekrar (İng. heptad repeat) olarak adlandırılan çok karakteristik bir dizi motifi içerirler, bu motif dizi boyunca kendini her yedi kalıntıda bir tekrar eder. Birinci ve dördüncü kalıntılar (a ve d pozisyonları olarak bilinir) hemen hep hidrofobiktir, (dördüncü kalıntı tipik olarak lösindir) ve sarmal demetinin içinde beraber istif olurlar. Genelde 5. ve 7. kalıntılar (e ve g pozisyonları) zıt yüklüdür ve elektrostatik etkileşimlerle stabilize olan bir tuz köprüsü oluştururlar. Keratin ve miyozin gibi fibröz proteinler ve bazı başka dimerleşen proteinler de, genelde sarımlı sarım yapılar oluşturur. Bir çift sarımlı sarım -dört sarmal demeti- proteinlerde çok sık görülen bir yapısal motiftir. Örneğin, insan büyüme hormonunda ve birkaç sitokrom çeşidinde mevcuttur. Bakterilerde plazmit ikileşmesini sağlayan Rop proteini ilginç bir vakadır, bir polipeptit bir sarımlı sarım oluşturur ve iki monomer onunla bir araya gelip bir dört-sarmal demeti oluşturur.
Bir sarmalı oluşturan amino asitler bir sarmal teker üzerinde çizilebilir, bu gösterim sarmalı oluşturan amino asit kalıntılarının doğrultularını resimler. Globuler proteinlerde, ve ayrıca sarımlı sarım ve lösin fermuarı gibi özelleşmiş yapılarda, bir alfa sarmal iki "yüz" sergiler: biri proteinin içine yönelik başlıca hidrofobik amino asitlerden oluşmuştur, öbürü ise proteinin çözeltiye temas eden yüzeyinde yer alan polar amino asitlerden oluşur.
Fonksiyonel roller
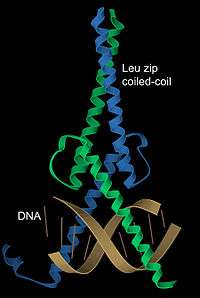
DNA'ya bağlanma
α-sarmalların DNA'ya bağlanan motifler için, sarmal-dönüş-sarmal, lösin fermuar ve çinko parmak motifleri dahil olmak üzere, özel bir önemi vardır. 1,2 nanometre olan α sarmal çapının, B-biçimli DNA'nın büyük oyuğunun genişliğine eşit olması, gayet uygun bir yapısal özellik yaratır. Ayrıca, sarımlı sarım (veya lösin fermuar) dimerleri kolaylıkla DNA'da sık görülen simetrik tekrarlı dizilerle temas kurabilir.[19] Bunun bir örneği transkripsiyon faktörü Max'tır (soldaki resme bakınız), bu protein sarımlı sarım kullanarak dimerleşir ve diğer bir sarmal çiftini DNA büyük oyuğu ile etkileşmek üzere uygun konuma getirir.
Membran aşımı
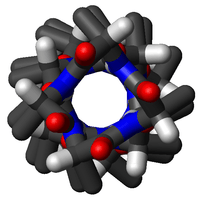
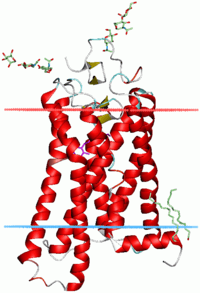
α-sarmallar biyolojik membranları içinden geçen en yaygın protein yapı elemanıdır.[20] Bunun nedeni, sarmal yapının, omurgadaki tüm hidrojen bağlarının dahilî olarak oluşmasını ve, eğer yan zincirler hidrofobikse, membran ile temas halinde hiçbir polar grup kalmamasını, sağlamasıdır. Proteinler bazen tek, bazen bir çift, en klasik olarak da yukarı-aşağı inip çıkan ve bir halka şeklinde dizilmiş yedi tane membran aşan sarmal ile membrana tutturulmuş olur. Yedi sarmallı membran aşan proteinlerin en bilinen örnekleri rodopsinler (sağdaki resme bakınız) ve G-protein-kenetli reseptörlerdir.
Mekanik özellikler
Eksen boyunca gerilme deformasyonu, α-sarmal zengini çoğu lifte ve dokularda olur, bunun sonucu üç fazlı bir sert-yumuşak-sert teğet zorlanım çarpanı (İng. tangent modulus) gösterir.[21] Faz I, sarmalın homojen olarak gerildiği bir küçük deformasyon safhasıdır, bunun ardından gelen Faz II'de H-bağlarının kopmasıyla alfa-sarmal dönmeleri bozulur. Faz III, tipik olarak kovalent bağ gerilimiyle ilişkili büyük ölçekli bir deformasyondur.
Dinamik özellikler
Raman spektroskopisi ile incelenip kontinuum-benzeri bir model ile analiz edilince, proteinlerdeki bazı alfa sarmalların düşük frekanslı, akordeon-vari hareket sergilediği görülebilir.[22][23][24]
Sarmal-sarım geçişi
Amino asitlerin homopolimerleri (poli-lizin gibi) düşük sıcaklıkta bir α-sarmal yapılı olup, yüksek sıcaklıklarda "eriyen" bir yapı gösterebilirler. Bu sarmal-sarım geçiş (İng. helix-coil transition) bir zamanlar protein denatürasyonuna benzer bir süreç olarak düşünülürdü. Bu geçişin istatistik mekanik analizi iki parametreye bağlı bir transfer matris yöntemi ile modellenebilir: bunlar sarmal oluşturma eğilimi ve sarmalı uzatma eğilimi.
Sanatta α-sarmal

Julian Voss-Andreae Almanya doğumlu bir heykeltıraş olup deneysel fizik ve heykeltıraşlık dallarında diplomaları vardır. 2001'den beri Voss-Andrea "protein heykeller" yaratmaktadır, α-sarmal onun en tercih ettiği cisimdir.[25] Voss-Andreae bambu ve tüm ağaç gibi çeşitli malzemelerle α-sarmallar yapmıştır. α-sarmalın kaşifi Linus Pauling'in anısına Voss-Andrea'nın 2004'te yarattığı bir anıt, α-sarmal şeklinde biçimlendirilmiş bir çelik kirişten yapılmıştır. 3 metre yüksekliğindeki parlak kırmızı heykel Pauling'in Portland, Oregon'daki çocukluk evinin önünde durmaktadır.
Ayrıca bakınız
- Katlanma (kimya)
- Beta yaprak
- Oyuk içine yumru istifleme
- İkincil yapı
- Üçüncül yapı
- Davydov soliton
Kaynakça
- ↑ Kendrew, JC; Dickerson, RE; Strandberg, BE; Hart, RG; Davies, DR; Phillips, DC; Shore, VC (1960). "Structure of myoglobin: A three-dimensional Fourier synthesis at 2 Å resolution". Nature 185 (4711): 422–427. DOI:10.1038/185422a0. PMID 18990802.
- ↑ Neurath, H (1940). "Intramolecular folding of polypeptide chains in relation to protein structure". Journal of Physical Chemistry 44: 296–305. DOI:10.1021/j150399a003.
- ↑ Taylor, HS (1942). "Large molecules through atomic spectacles". Proceedings of the American Philosophical Society 85: 1–12.
- ↑ Huggins, M (1943). "The structure of fibrous proteins". Chemical Reviews 32: 195–218. DOI:10.1021/cr60102a002.
- ↑ Bragg, WL; Kendrew JC, Perutz MF (1950). "Polypeptide chain configurations in crystalline proteins". Proceedings of the Royal Society a 203: 321–?. DOI:10.1098/rspa.1950.0142.
- ↑ Pauling, L; Corey RB, Branson HR (1951). "The Structure of Proteins: Two Hydrogen-Bonded Helical Configurations of the Polypeptide Chain". Proceedings of the National Academy of Science in Washington 37 (4): 205–211. DOI:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1063337.
- ↑
- ↑ IUPAC-IUB Commission on Biochemical Nomenclature (1970). "Abbreviations and symbols for the description of the conformation of polypeptide chains". Journal of Biological Chemistry 245: 6489–6497. http://www.chem.qmul.ac.uk/iupac/misc/ppep1.html.
- ↑ Kabsch, K; Sander C (1983). "Identification of structural motifs from protein coordinate data: secondary structure and first-level supersecondary structure". Biopolymers 22 (12): 2577–2637. DOI:10.1002/bip.360221211. PMID 6667333.
- 1 2 Richardson, JS (1981). "The Anatomy and Taxonomy of Proteins". Advances in Protein Chemistry 34: 167–339 . DOI:10.1016/S0065-3233(08)60520-3. PMID 7020376.
- ↑ Lovell SC et al. (2003). "Structure validation by Cα geometry: φ,ψ and Cβ deviation". Proteins 50 (3): 437–450. DOI:10.1002/prot.10286. PMID 12557186.
- ↑ Dickerson, RE; Geis I (1969). Structure and Action of Proteins. Harper, New York.
- ↑ Schiffer M, Edmundson AB (March 1967). "Use of helical wheels to represent the structures of proteins and to identify segments with helical potential". Biophys. J. 7 (2): 121–35. DOI:10.1016/S0006-3495(67)86579-2. PMC 1368002. PMID 6048867. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1368002.
- 1 2 Chou KC, Zhang CT, Maggiora GM (May 1997). "Disposition of amphiphilic helices in heteropolar environments". Proteins 28 (1): 99–108. DOI:10.1002/(SICI)1097-0134(199705)28:1<99::AID-PROT10>3.0.CO;2-C. PMID 9144795.
- ↑ Kurochkina N (May 2010). "Helix-helix interactions and their impact on protein motifs and assemblies". J. Theor. Biol. 264 (2): 585–92. DOI:10.1016/j.jtbi.2010.02.026. PMID 20202472.
- ↑ Hudgins, RR; Jarrold MF (1999). "Helix Formation in Unsolvated Alanine-Based Peptides: Helical Monomers and Helical Dimers". Journal of the American Chemical Society 121: 3494–3501. DOI:10.1021/ja983996a.
- ↑ Kutchukian, PS; Yang JS, Verdine GL, Shakhnovich EI (2009). "All-Atom Model for Stabilization of alpha-Helical Structure in Peptides by Hydrocarbon Staples". Journal of the American Chemical Society 131 (13): 4622–4627. DOI:10.1021/ja805037p. PMC 2735086. PMID 19334772. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2735086.
- ↑ Pace CN, Scholtz JM (July 1998). "A helix propensity scale based on experimental studies of peptides and proteins". Biophys. J. 75 (1): 422–7. DOI:10.1016/S0006-3495(98)77529-0. PMC 1299714. PMID 9649402. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1299714.
- ↑ Branden & Tooze, chapter 10
- ↑ Branden & Tooze, chapter 12
- ↑ T. Ackbarow, X. Chen, S. Keten, M.J. Buehler (2007). "Hierarchies, multiple energy barriers and robustness govern the fracture mechanics of alpha-helical and beta-sheet protein domains". PNAS 104 (42): 16410–16415. DOI:10.1073/pnas.0705759104. PMC 2034213. PMID 17925444. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2034213.
- ↑ Painter PC, Mosher LE, Rhoads C (July 1982). "Low-frequency modes in the Raman spectra of proteins". Biopolymers 21 (7): 1469–72. DOI:10.1002/bip.360210715. PMID 7115900.
- ↑ Chou KC (December 1983). "Identification of low-frequency modes in protein molecules". Biochem. J. 215 (3): 465–9. PMC 1152424. PMID 6362659. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1152424.
- ↑ Chou KC (May 1984). "Biological functions of low-frequency vibrations (phonons). III. Helical structures and microenvironment". Biophys. J. 45 (5): 881–9. DOI:10.1016/S0006-3495(84)84234-4. PMC 1434967. PMID 6428481. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1434967.
- ↑ Voss-Andreae, J (2005). "Protein Sculptures: Life's Building Blocks Inspire Art". Leonardo 38: 41–45. DOI:10.1162/leon.2005.38.1.41.
Dış bağlantılar
- NetSurfP - Secondary Structure and Surface Accessibility predictor (İkincil yapı ve yüzey erişilirlik öngörücüsü)
- bir α-sarmalın enteraktif modeli
- α-sarmalın animasyonlu ayrıntıları
- Artist Julie Newdoll'un Web sitesi
- Artist Julian Voss-Andreae'un Web sitesi